实验室小白遇上引物设计,一片迷茫,不知所措。
师姐:现在常用的的引物设计软件有Oligo、PrimerPremier、DNA man等等,还有在线引物设计软件。咱实验室的软件给你拿去。
(师姐比师兄靠谱,真的是手把手的教了,边操作边讲解。)
1. 在NCBI上搜索到该基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2. PrimerPremier5.0安装上。
3. 打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。点击Primer,进入引物窗口。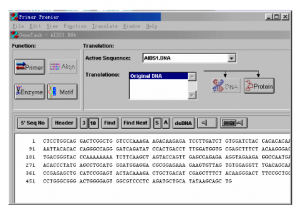
4. 此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。在Search Parameters里面,可以设定相应参数。一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp。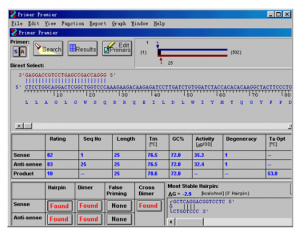
5. 点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。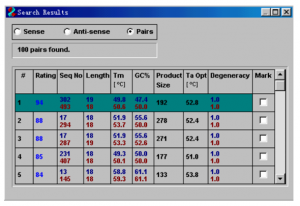
6. 对于引物的序列,可以简单查看一下,避免出现下列情况:3’端不要以A结尾,最好是G或者C,T也可以;3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。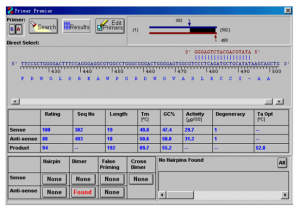
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。
7. 在Primer5窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择File-Print-Current pair,使用PDF虚拟打印机,即可转换为Pdf文档,里面有该引物的详细信息。
8. 当然,引物确定后,对于上游和下游引物分别进行Blast分析,一般来说,多少都会找到一些其他基因的同源序列,此时,可以对上游引物和下游引物的blast结果进行对比分析,只要没有交叉的其他基因的同源序列就可以。
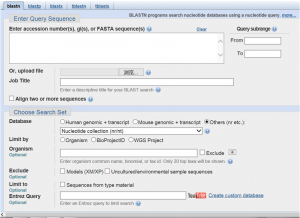
(1)选择blast中的nucleotide blast
(2)在choose search set中,database根据与操作基因的种属选择human,mouse或others。在program selection中选择somewhat similar sequences 项,点击blast按钮。
(3)等待几秒后,出现显示blast结果的网页。线段颜色与上面分值越高,特异性越好。(两条线段间有连线的代表这些序列与上游引物匹配,并与下游引物互补,理论上可以扩增基因片段。没有连线的表示单条引物与该基因一致。)
(4)点击线段跳转到该基因的结果信息概要:
(Accession:数据库系列的身份证,点击后可以获得该序列的信息
Dessecription:系列的简单描述。
Total score:高的就是两线段间有连线的
E value:被比对的两个序列不相关的可能性。E值越低越有意义。)
(5)一般能在结果找到你的目的基因的引物正确性都没问题。
(如果你找到一大批同物种的不同基因,而且分数也很高,说明引物设计的特异性不高,极易出现非特异性扩增。)
好了,今天就分享到这里,PCR相关实验做了几百遍,区区引物设计小case,觉得师姐的技巧实用的可以转发朋友圈或发给你的朋友哦,好东西要分享。想持续获得免费生物资讯,可以关注我的微信号,我们一起畅聊实验室里的那些事儿。
扫描右侧二维码,关注英格恩公众号,随时阅读沟通。
 英格恩生物技术博客 生物实验干货分享
英格恩生物技术博客 生物实验干货分享